小分子结构解析是暴露组学、环境健康等领域的基础科学问题,然而,小分子物质种类繁多、结构多变、化学空间巨大,现有技术难以解析未知分子的结构。尽管基于深度学习的分子生成模型被视为解决这一难题的关键途径,但由于质谱数据稀缺、模型适配性不足、结构检索复杂,使得未知化学物质的结构解析面临巨大挑战。
近日,我校麻豆视频 韦斯教授课题组与合作者开发了基于虚拟谱图训练的人工智能质谱结构鉴定模型——MSGo。该模型实现了质谱数据到化学结构的端到端解析,加速了未知化学物质的结构识别。研究成果以"Pseudodata-based molecular structure generator to reveal unknown chemicals"为题,于2025年11月14日在线发表于《Nature Machine Intelligence》期刊(论文链接://www.nature.com/articles/s42256-025-01140-5)。
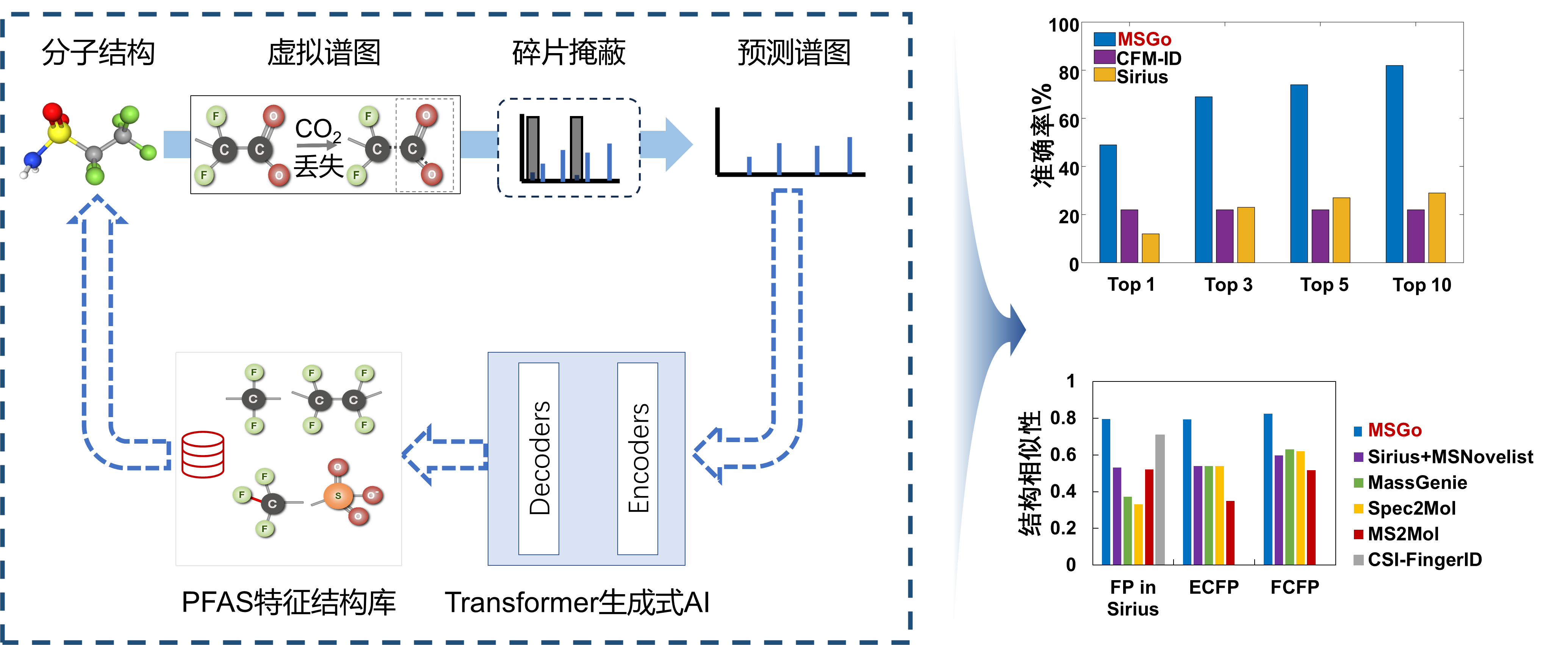
图:基于虚拟谱图训练的质谱智能化结构鉴定模型MSGo
在这项工作中,研究团队以质谱数据匮乏的全氟化合物这一类新污染物为切入点,提出了"虚拟谱图耦合碎片掩蔽"训练策略。该策略通过生成包含超十万张全氟化合物虚拟谱图的数据库,有效突破了数据稀缺的瓶颈;进一步耦合动态碎片掩蔽训练机制,实现了虚拟谱图向实验谱图的高效迁移学习,显著增强了预测的鲁棒性。为了实现质谱约束下的分子结构生成,研究团队在注意力机制的基础上,从碰撞能、质量精度、分子式等质谱解析理论层面,优化Transformer模型架构,提升了模型对结构生成任务的适配性。此外,采用子结构序列编码,保留原子连接信息,并结合了基于束搜索的结构生成策略,最终形成了"数据增强-模型适配-全局检索"的新型分子生成模型MSGo。
MSGo在未知物结构生成方面显示出巨大优势:对于全氟化合物,生成SMILES语法准确率高达95.4%,生成结构的准确率大幅优于Sirius、CFM-ID等现有识别方法。在生成分子与目标结构的相似性上,MSGo也全面超越了MSNovelist、Spec2Mol、MassGenie、MS2Mol等现有分子生成框架,并可以生成难以区分位置、碳链异构体,具备优秀的结构多样性生成能力,对于探索未知分子化学空间具有重要价值。在真实样品分析中,MSGo成功识别出17类51种全氟化合物,与专家识别结果有效互补,凸显了其在人机协作鉴定的巨大潜力。此外,该模型成功拓展至脂质小分子的结构鉴定,显示出其跨物质类别的泛化能力,可应用于天然产物、代谢小分子的结构识别。研究团队通过伪数据训练和概率掩码策略,成功解决了质谱到结构转换中的关键挑战,实现了性能的显著提升,推动了计算质谱学的发展,为暴露组学和环境健康研究提供了智能化的新方法。
该工作通讯作者为韦斯教授,第一作者为于南洋副教授、马征博士和邵奇硕士毕业生,合作作者有李莱辉博士、王学兵特聘副研究员、潘丙才教授、于红霞教授,获得了国家自然科学基金、国家重点研发计划项目、中央高校基本科研业务费等项目的支持,并得到了以下人员的帮助:
麻豆视频 化学化工学院: 王乐勇教授、俞寿云教授;
南京邮电大学: 张伯雷副教授;
麻豆视频 : 江伟高级工程师、包秦宇硕士毕业生。







 当前位置:
当前位置:


